「Nmny: step by step」の版間の差分
ページの作成:「== nmny: step by step == nmny の最新版は http://mux.eee.u-ryukyu.ac.jp/nmny/trial で公開されています。WebGLの有効化手順を参照の上、ブラ...」 |
編集の要約なし |
||
| 90行目: | 90行目: | ||
<br clear="both"> | <br clear="both"> | ||
== アンカー表示画面の操作 == | |||
=== 配列の表示位置を手動で調整する === | |||
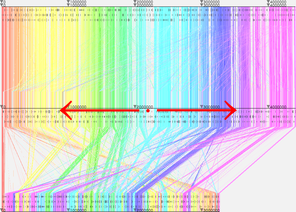
下の図のように各ゲノムに対応するグレーの帯をドラッグすることで、左右に移動することができます。何も表示されていないところをドラッグした場合には、表示されている画像全体を移動できます。 | |||
[[Image:mainscreen-move.png|296px|border]] [[Image:mainscreen-move2.png|339px|border]] | |||
=== 配列の表示位置をアンカーによって調整する === | |||
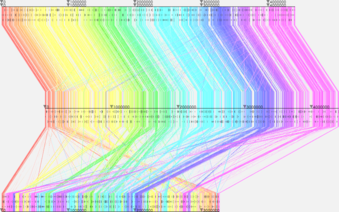
配列の位置は手動で調整するだけでなく、アンカーを用いて自動で調整することもできます。下の図の例のように、配列の上下の、アノテーション情報のマーカーが表示されていない部分をクリックすることで、そこを通っているアンカーを用いて配列の表示位置が調整されます。 | |||
[[Image:mainscreen-align.png|296px|border]] [[Image:mainscreen-align2.png|437px|border]] | |||
2014年7月14日 (月) 08:27時点における最新版
nmny: step by step
nmny の最新版は http://mux.eee.u-ryukyu.ac.jp/nmny/trial で公開されています。WebGLの有効化手順を参照の上、ブラウザの設定を済ませてからアクセスしてください。
データセットを選ぶ
初期画面は以下のようになります。
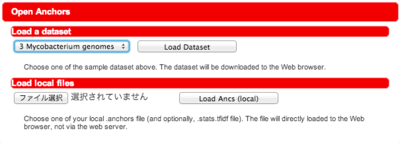
"Load a dataset" からは、サーバ上にある Murasaki の出力を選択することができます。nmny の公開サーバでは現在、いくつかのサンプルを公開していますが、将来的にはサンプル数を増やしたり、ユーザ個人のデータを登録できるようにする計画です。
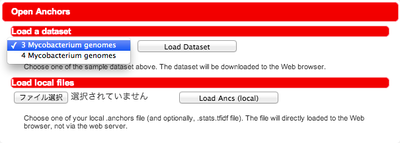
File API に対応しているブラウザの場合は、"Load local files" も表示されます。これを使うと、ユーザのコンピュータ上でMurasakiが出力した ".anchors" ファイルと、".anchors.stats.tfidf" ファイル (後者はオプション) を選択し、nmny に読み込むこともできます。
基本画面
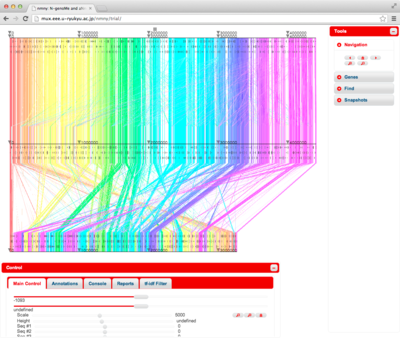
nmny が起動すると以下のような画面になります。アライメントは左上から描画され、左下と右端に、操作のためのウィンドウが配置されます。アライメントはマウス操作により移動することができ、ふたつのウインドウはそれぞれサイズ変更や、アイコン化を行うことができます (アライメントの描画操作については後のセクションでくわしく説明します)。
横方向のグレーの直線は、Murasaki に読み込まれた各ゲノムを表しており、この例では3つのゲノム配列が読み込まれています。ゲノム上での位置は、目盛りの数字で表示されます。アノテーション情報が読み込まれている場合には、この例のように、ゲノムを表す線の下 (一番下に表示される配列については上) に、アノテーション情報を表すグレーのマーカーが表示されます。この例ではゲノム全体を表示しているため、マーカーは非常に小さくなっていますが、拡大すれば遺伝子の方向なども判別できるような表示になります。デフォルトでは、ひとつのアノテーショントラックは3段に分割して表示されますが、この段数は "Control" ウィンドウの "Annotation" タブで変更することができます。
縦・斜め方向の着色されている線はMurasakiによって出力されたアンカーで、色は (現在のところ) 一番上に表示されているゲノム上の位置によって決まります。
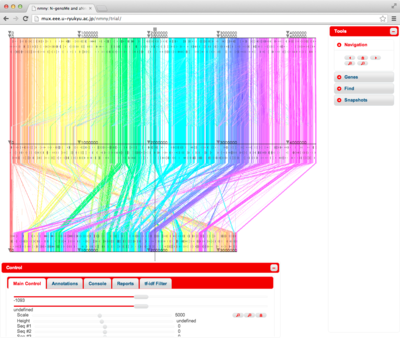
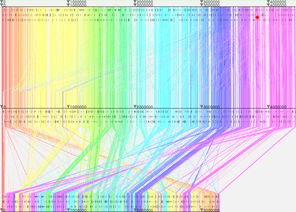
また、この図の 2Mbp 付近の画面上部にグレーの四角が表示されていますが、これはカーソル位置を示すもので、マウスカーソルを重ねると下の例のようにカーソルが表示され、ドラッグにより移動することができます。カーソルは、後述するズームイン・ズームアウトの操作で使用されます。
Tools ウィンドウ
画面右側の "tools" ウィンドウを使ってさまざまな操作を行うことができます。
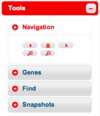
"Navigation" タブでは、左右への移動、初期表示へのリセット、拡大縮小が可能です。
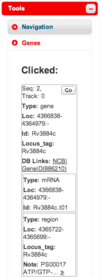
アノテーショントラック上のマーカーをクリックすると、対応するアノテーション情報が "Genes" タブに表示されます。また、アンカーで接続されている他の配列のアノテーショントラック上の情報が見つかれば、それも表示されます。
場合によってはこの情報は非常に長くなるので、スクロールして読む必要があるかもしれません。読みやすくするには、ウィンドウの幅を広げてやるとよいでしょう。

"Find" タブでは、遺伝子名などのキーワードで検索をすることができます。検索結果の "Go" ボタンを押すと、その部分が拡大表示されます。
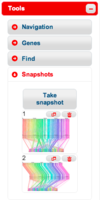
"Snapshots" タブでは表示されている画面のスナップショットを画像としてとっておくことが可能です。"Take snapshot" ボタンで現状の画面がタブに保存されます。保存した画像はフルサイズで表示したり、削除することもできます。
Control ウィンドウ
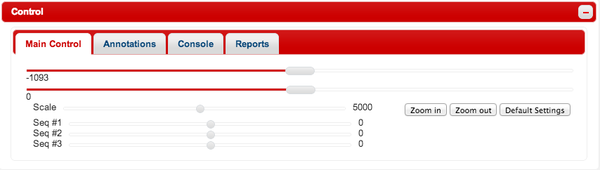
Control ウィンドウでは、nmny の表示をコントロールしたり、アノテーショントラックの追加などを行うことができます。初期状況で開いているのは "Main Control" タブ で、スライダーを使って上下左右の表示位置や、横方向のスケールを調整することができます。また、配列毎に左右に動かすこともできます。
"Zoom in" および "Zoom out" のボタンを使うと、カーソル位置を中心に横方向のスケールが変更できます。スケールや表示位置を元に戻すには、"Default Settings" のボタンを使用します。
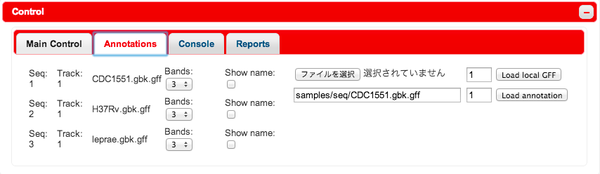
"Annotations" タブではアノテーショントラックの表示の調整と、追加を行います。すでに読み込まれているトラックは左側に表示され、"Bands" ではアノテーショントラックを何段に分けて表示するかを、"Show name" では、遺伝子名などの表示・非表示を切り替えることができます。
また、右側の "Load local GFF" や "Load annotation" を使うと、自分の持っているGFFファイルを読み込んだり、サーバ上のGFFファイルを利用することができます。
"Console" タブにはデバッグ情報が出力されます。開発者向けです。
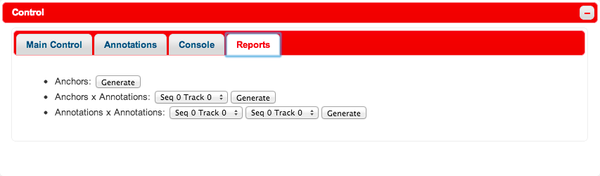
"Reports" では、アンカーを用いてアノテーションの相互参照リストなどを作成することができます。
- Anchors: アンカーのリストを出力します。
- Anchors x Annotations: アノテーションが付与されている領域中のアンカーの数などを出力します。
- Annotations x Annotations: アンカーで接続されたアノテーション情報のリストを出力します。
レポートはファイルに保存することもできます。
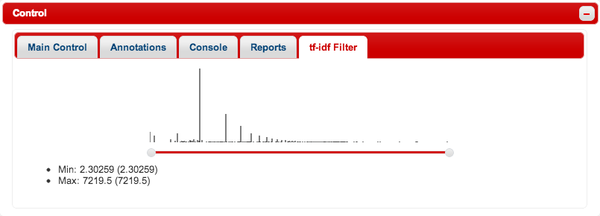
Murasaki の tf-idf スコアが読み込まれている場合には "tf-idf Filter" タブが表示されます。このタブに表示されるスコアのヒストグラムの下のスライダーで、表示するスコアの範囲を設定することができます。
アンカー表示画面の操作
配列の表示位置を手動で調整する
下の図のように各ゲノムに対応するグレーの帯をドラッグすることで、左右に移動することができます。何も表示されていないところをドラッグした場合には、表示されている画像全体を移動できます。
配列の表示位置をアンカーによって調整する
配列の位置は手動で調整するだけでなく、アンカーを用いて自動で調整することもできます。下の図の例のように、配列の上下の、アノテーション情報のマーカーが表示されていない部分をクリックすることで、そこを通っているアンカーを用いて配列の表示位置が調整されます。