「メインページ」の版間の差分
ナビゲーションに移動
検索に移動
編集の要約なし |
編集の要約なし |
||
| 3行目: | 3行目: | ||
== nmny とは == | == nmny とは == | ||
nmny (んむにー) は、 gmv の後継版で、Web | nmny (んむにー) は、[http://murasaki.dna.bio.keio.ac.jp/gmv.html GMV] の後継版で、Web ブラウザ上で動作する比較ゲノムブラウザです。主に[http://murasaki.dna.bio.keio.ac.jp/ Murasaki]による比較ゲノム解析の結果を可視化することを目的に作られており、アライメント結果とアノテーション情報を重ねて表示したりすることができます。GMV は[http://www.gtk.org/ GTK+]をベースにしたデスクトップアプリケーションですが、nmny では web サービスでの使用を考えて、ブラウザ上で HTML5 + WebGL による描画を行います (詳しい能書きは[[nmnyの特徴]]をご覧ください。) | ||
nmny の動作には WebGL に対応したブラウザが必要です。各ブラウザで WebGL を有効化するための設定は[[WebGLの有効化手順]]をお読みください。 | |||
== nmny: step by step == | |||
nmny の最新版は http://mux.eee.u-ryukyu.ac.jp/nmny/trial で公開されています。[[WebGLの有効化手順]]を参照の上、ブラウザの設定を済ませてからアクセスしてください。 | |||
=== データセットを選ぶ === | |||
初期画面は以下のようになります。 | |||
[[Image:startup1.png|center]] | |||
"Load a dataset" からは、サーバ上にある Murasaki の出力を選択することができます。 | |||
[[Image:startup2.png|center]] | |||
File API に対応しているブラウザの場合は、"Load local files" も表示されます。ここで、Murasakiが出力した ".anchors" ファイルと、".anchors.stats.tfidf" ファイル (後者はオプション) を選択して nmny に読み込むこともできます。 | |||
=== 基本画面 === | |||
[[Image:screen-basic.png|center|400px]] | |||
2013年3月6日 (水) 05:58時点における版
nmny とは
nmny (んむにー) は、GMV の後継版で、Web ブラウザ上で動作する比較ゲノムブラウザです。主にMurasakiによる比較ゲノム解析の結果を可視化することを目的に作られており、アライメント結果とアノテーション情報を重ねて表示したりすることができます。GMV はGTK+をベースにしたデスクトップアプリケーションですが、nmny では web サービスでの使用を考えて、ブラウザ上で HTML5 + WebGL による描画を行います (詳しい能書きはnmnyの特徴をご覧ください。)
nmny の動作には WebGL に対応したブラウザが必要です。各ブラウザで WebGL を有効化するための設定はWebGLの有効化手順をお読みください。
nmny: step by step
nmny の最新版は http://mux.eee.u-ryukyu.ac.jp/nmny/trial で公開されています。WebGLの有効化手順を参照の上、ブラウザの設定を済ませてからアクセスしてください。
データセットを選ぶ
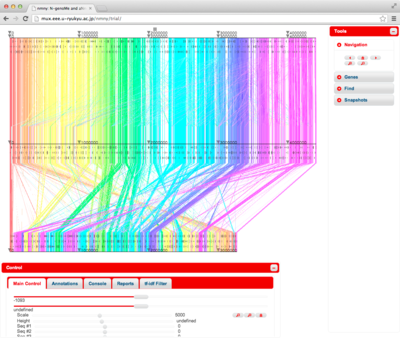
初期画面は以下のようになります。
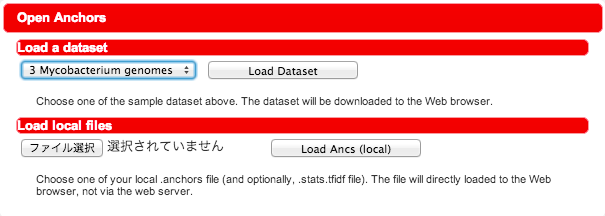
"Load a dataset" からは、サーバ上にある Murasaki の出力を選択することができます。
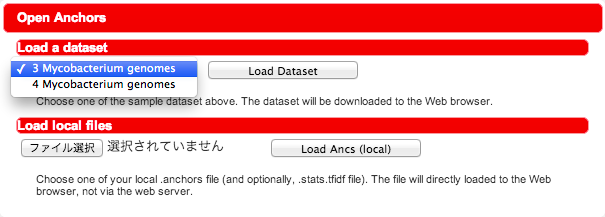
File API に対応しているブラウザの場合は、"Load local files" も表示されます。ここで、Murasakiが出力した ".anchors" ファイルと、".anchors.stats.tfidf" ファイル (後者はオプション) を選択して nmny に読み込むこともできます。
基本画面